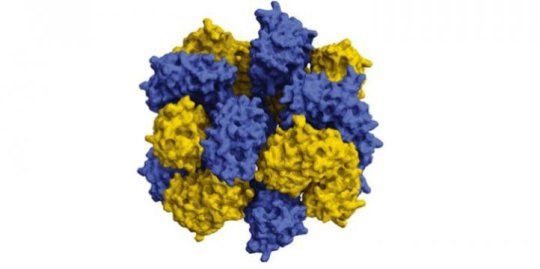

科學家利用從水母中提取的蛋白質組裝了一個複雜的16種蛋白質結構,該結構由兩種疊加的八肽組成。這項研究可應用於有用的技術,如藥物靶向,人工能源收集,「智能」感測和建築材料,等等。通過XSEDE分配對斯坦胡枝子2(TACC)和彗星(SDSC)的結構精細測量的計算模型。
紅細胞太神奇了。它們從我們的肺裡吸走氧氣,把氧氣帶到我們的全身,以維持我們的生命。紅細胞中的血紅蛋白分子通過改變它的形狀來輸送氧氣。血紅蛋白中有四個相同的蛋白拷貝,開著和合著,就像花瓣一樣,在結構上相互耦合,相互響應。利用超級計算機,科學家們才剛剛開始設計能自我組裝的蛋白質,使它們結合在一起,就像血紅蛋白這樣的給人生命的分子。科學家們說,他們的方法可以應用於有用的技術,如藥物靶向、人工能源收集、「智能」感知和建築材料等等。
一個科學小組通過增壓蛋白質來完成這項工作,這意味著他們改變了蛋白質的亞基,即氨基酸,從而人為地給予蛋白質一個高正負電荷。科學家們利用從水母中提取的蛋白質,通過增壓組裝了一個複雜的16種蛋白質結構,由兩種堆積的八肽組成,這一發現發表在2019年1月的「華爾街日報」(The Journal)上。自然化學.
然後,研究小組使用超級計算機模擬來驗證和告知這些實驗結果。在德克薩斯高級計算中心(TACC)和聖地亞哥超級計算機中心(SDSC)上對斯坦胡枝子2的超級計算機撥款是通過XSEDE授予研究人員的,XSEDE是由國家科學基金會(NSF)資助的極限科學和工程探索環境(ExtremeScience And Engineering Discovering Environment)。
「我們發現,通過獲取通常不相互作用的蛋白質,我們可以複製出高正電或高負電荷的拷貝,」聯合作者安娜·西蒙(AnnaSimon)說,她是奧斯汀大學埃靈頓實驗室(Ellington Lab)的博士後研究員。西蒙說:「結合高正負性的拷貝,我們可以將蛋白質組裝成非常特殊的結構組裝。」科學家們稱他們的策略為「超荷蛋白質組裝」,通過結合工程超荷變體來驅動確定的蛋白質相互作用。
研究的合著者詹斯·格拉澤補充道:「我們利用了自然界中一個非常著名的基本原理,即相反的電荷吸引人。」格拉澤是密歇根大學化學工程系Glotzer集團的助理研究科學家。「安娜西蒙的小組發現,當他們混合這些帶電的綠色熒光蛋白變體,他們得到高度有序的結構。這是一個真正的驚喜,」格拉澤說。
堆疊的八進位結構看起來像一個編織的戒指。它由16種蛋白質組成-兩個相互纏繞的八個環,它們在非常特殊的、離散的斑塊中相互作用。西蒙解釋說:「之所以如此難以合成相互作用的蛋白質,是因為製作這些相互作用的斑塊,並讓它們都對齊,使它們能夠組裝成更大、更規則的結構,這是非常困難的。」他們通過在綠色熒光蛋白(Gfp)中添加許多正負電荷來解決這個問題,綠色熒光蛋白是一種經過充分研究的「實驗室老鼠」蛋白,來源於黃花水母。
帶正電荷的蛋白質,他們稱之為藍色熒光蛋白(Ceru)+32,有更多的機會與帶負電荷的GFP-17相互作用。西蒙說:「通過給予這些蛋白質所有這些機會,這些不同的地方它們可能相互作用,它們就能夠選擇合適的蛋白質。」「有一些模式和相互作用是存在的,它們是可用的,而且受到了積極的青睞,我們不一定事先預測它們就能組裝成這些特定的形狀。」
為了獲得基因工程的熒光蛋白,Simon和他的合著者Arti Postukuchy、Jimmy Gollihar和Barrett Morrow對他們的基因進行了編碼,其中包括一種化學標記,用於純化可攜帶的DNA片段,即大腸桿菌中的質粒,然後獲取大腸桿菌生長的標記蛋白。科學家們把這些蛋白質混合在一起。他們最初認為這些蛋白質可能只是相互作用,形成了結構不規則的大塊狀。「但是,我們一直看到的是這個奇怪的,有趣的12納米左右的峰值,它比一大團蛋白質小得多,但比單一的蛋白質要大得多,」西蒙說。
他們測量了奧斯丁德州材料研究所的Zetasizer儀器所形成的粒子的大小,並證實了這些粒子同時含有金屬色和綠色熒光蛋白F rster Resonance Energy Transfer(FRET),後者測量不同顏色熒光蛋白之間的能量傳遞,以響應不同的光能量,以確定它們是否緊密相連。負染色電子顯微鏡鑒定了粒子的特殊結構,由UT奧斯汀的分子生物科學助理教授DavidTaylor小組進行。結果表明,12 nm粒子由16種蛋白質組成。西蒙說:「我們發現它們是這些形狀優美的花狀結構。」來自奧斯汀大學泰勒小組的合著者周一舟(音譯)進一步提高了解析度,使用低溫電子顯微鏡來揭示堆疊的八面體的原子級細節。
根據jensgaser的說法,計算模型細化了蛋白質如何排列成美麗的花朵狀結構的清晰圖像的測量結果。「我們必須想出一個足夠複雜的模型來描述帶電綠色熒光蛋白的物理特性,並展示所有相關的原子化細節,但它的效率足以讓我們在一個真實的時間尺度上模擬這個過程。」有了一個完全原子化的模型,我們就需要一年多的時間才能從計算機上得到一個模擬結果,不管計算機的速度有多快,「格拉澤說。
他們在不犧牲蛋白質相互作用的重要細節的情況下,通過降低解析度來簡化模型。「這就是為什麼我們使用一個模型,在這個模型中,蛋白質的形狀精確地由分子表面來表示,就像從蛋白質的晶體結構中測量出來的那樣,」格拉澤補充道。
密西根大學化學工程研究生Vyas Ramasubramani說:「真正幫助我們扭轉這一局面並改善我們能夠從模擬中獲得的是冷凍-EM數據。」Ramasubramani說:「這確實幫助我們找到了最優的結構,從而幫助我們驗證了我們正在提出的穩定性論點,並希望未來能夠預測我們可以破壞或修改這種結構的方式。」
科學家們需要大量的計算能力來進行他們想要的規模的計算。
Ramasubramani說:「我們使用XSEDE基本上是以這些巨大的系統為例,在這些系統中,有許多不同的部分相互互動,並同時計算所有這些,以便當您開始將系統向前移動時,您可以了解到它將如何在某種真實的時間尺度上進化,」Ramasubramani說。「如果你試著做和我們在筆電電腦上做的相同的模擬,你需要幾個月甚至幾年的時間才能真正理解某種結構是否穩定。」對於我們來說,如果不能使用XSEDE,你可以同時使用48個核心,48個計算單元來使這些計算高度並行,我們就會這樣做得慢得多。「
TACC中的斯坦胡枝子2超級計算機包含4,200個英特爾騎士登陸和1,736個Intel Skylake X計算節點。每個Skylake節點有48個核,是計算機處理器的基本單元。「斯坦胡枝子2超級計算機的Skylake節點有助於實現計算這些靜電相互作用所必需的性能,這些靜電相互作用以有效的方式作用於相對帶電的蛋白質之間,」格拉澤說。「斯坦胡枝子2超級計算機的可用性正好是我們進行這些模擬的最佳時機。」
最初,科學小組在SDSC的彗星系統上測試了他們的模擬。Ramasubramani說:「當我們第一次弄清楚該使用什麼樣的模型,以及這個簡化的模型是否能給我們提供合理的結果時,彗星是一個很好的地方來嘗試這些模擬,」Ramasubramani說。「彗星是我們所做的偉大實驗。」
從更大的科學角度來看,科學家們希望這項工作能提高人們對自然界中為什麼如此多的蛋白質會齊聚,或結合在一起,形成更複雜和有趣的結構的理解。
西蒙說:「我們證明,不需要有一套非常具體、預先區分的計劃和互動來形成這些結構。」「這一點很重要,因為這意味著,我們很可能也很有可能利用我們想要製造齊聚的其他分子,同時產生帶正電和帶負電的變體,將它們結合起來,並為它們建立特定的有序結構。」
像骨頭、羽毛和貝殼這樣的天然生物材料可以是堅硬的,但重量輕。西蒙說:「我們認為超荷蛋白質組裝是一種更容易開發出具有令人興奮的合成性能的材料的方法,而不必花那麼多時間,也不必事先確切地知道它們將如何聚在一起。」「我們認為,這將加快合成材料的工程能力,並有助於這些納米結構蛋白材料的發現和探索。」
TAG: |